Você provavelmente conhece o pacote ggplot2, mas você sabia que também é possível criar gráficos animados sem nenhuma dificuldade? Essa é exatamente a função do pacote gganimate e hoje nós vamos conversar um pouco sobre as animações.
Ideia geral
O pacote gganimate parte de um objeto ggplot2 que pode ser decomposto em vários plots repetidos. A animação que vemos na tela é uma composição a partir de todas essas imagens. As possibilidades de composição são definidas pelas chamadas à funções transition_*, que definem de que modo a sua animação deve ser se comportar entre um gráfico e outro. Você pode conferir a nossa live ou o cheatsheet para ver alguns exemplos.
O exemplo abaixo usa transition_reveal(data_aplicacao) do pacote gganimate para transformar um ggplot2 comum em várias imagens que representam a curva de pessoas vacinadas ao longo do tempo:
library(tidyverse)
library(bigrquery)
conexao_covid <- dbConnect(
bigrquery::bigquery(),
project = "basedosdados",
dataset = "br_ms_vacinacao_covid19",
billing = "live-curso-r-bd-2"
)
conexao_populacao <- dbConnect(
bigrquery::bigquery(),
project = "basedosdados",
dataset = "br_ibge_populacao",
billing = "live-curso-r-bd-2"
)
vacinacao_base <- tbl(conexao_covid, "microdados_vacinacao") %>%
count(sigla_uf, data_aplicacao, dose) %>%
collect() %>%
mutate(
data_aplicacao = as.Date(data_aplicacao)
)
populacao_estados <- tbl(conexao_populacao, "municipio") %>%
filter(ano == 2020) %>%
collect() %>%
mutate(
id_estado = str_sub(id_municipio, 1, 2),
sigla_uf = case_when(
id_estado == "12" ~ "AC",
id_estado == "27" ~ "AL",
id_estado == "16" ~ "AP",
id_estado == "13" ~ "AM",
id_estado == "29" ~ "BA",
id_estado == "53" ~ "DF",
id_estado == "23" ~ "CE",
id_estado == "32" ~ "ES",
id_estado == "52" ~ "GO",
id_estado == "21" ~ "MA",
id_estado == "51" ~ "MT",
id_estado == "50" ~ "MS",
id_estado == "31" ~ "MG",
id_estado == "15" ~ "PA",
id_estado == "25" ~ "PB",
id_estado == "41" ~ "PR",
id_estado == "26" ~ "PE",
id_estado == "22" ~ "PI",
id_estado == "33" ~ "RJ",
id_estado == "24" ~ "RN",
id_estado == "43" ~ "RS",
id_estado == "11" ~ "RO",
id_estado == "14" ~ "RR",
id_estado == "42" ~ "SC",
id_estado == "35" ~ "SP",
id_estado == "28" ~ "SE",
id_estado == "17" ~ "TO"
)
) %>%
group_by(sigla_uf) %>%
summarise(
populacao = sum(populacao)
)
vacinacao_base_por_estado <- vacinacao_base %>%
ungroup() %>%
mutate(
regiao = case_when(
sigla_uf %in% c("SP", "RJ", "MG", "ES") ~ "Sudeste",
sigla_uf %in% c("SC", "PR", "RS") ~ "Sul",
sigla_uf %in% c("MT", "DF", "GO", "MS") ~ "Centro-Oeste",
sigla_uf %in% c("AC", "AM", "RO", "RR", "PA", "AP", "TO") ~ "Norte",
TRUE ~ "Nordeste"
)
) %>%
filter(dose == "2") %>%
group_by(regiao, sigla_uf) %>%
arrange(regiao, sigla_uf, data_aplicacao) %>%
mutate(
n_acum = cumsum(n),
) %>%
filter(data_aplicacao >= as.Date("2021-01-01")) %>%
left_join(
populacao_estados
) %>%
mutate(
percentual_vacinado = n_acum/populacao
)
populacao_regiao <- vacinacao_base_por_estado %>%
group_by(regiao) %>%
summarise(
populacao = sum(unique(populacao))
)
vacinacao_base_por_regiao <- vacinacao_base %>%
ungroup() %>%
mutate(
regiao = case_when(
sigla_uf %in% c("SP", "RJ", "MG", "ES") ~ "Sudeste",
sigla_uf %in% c("SC", "PR", "RS") ~ "Sul",
sigla_uf %in% c("MT", "DF", "GO", "MS") ~ "Centro-Oeste",
sigla_uf %in% c("AC", "AM", "RO", "RR", "PA", "AP", "TO") ~ "Norte",
TRUE ~ "Nordeste"
)
) %>%
filter(dose == "2") %>%
group_by(regiao, data_aplicacao) %>%
summarise(
n = sum(n)
) %>%
group_by(regiao) %>%
arrange(regiao, data_aplicacao) %>%
mutate(
n_acum = cumsum(n),
) %>%
filter(data_aplicacao >= as.Date("2021-02-01")) %>%
left_join(
populacao_regiao
) %>%
mutate(
percentual_vacinado = n_acum/populacao
)
library(gganimate)
grafico_por_regiao <- vacinacao_base_por_regiao %>%
#filter(regiao == "Sudeste") %>%
#filter(data_aplicacao <= as.Date("2021-04-15")) %>%
ungroup() %>%
arrange(data_aplicacao) %>%
#mutate(data_aplicacao = as.numeric(data_aplicacao)) %>%
ggplot(aes(x = data_aplicacao, y = percentual_vacinado, color = regiao)) +
#geom_col() +
#geom_line(size = 1.2) +
geom_point(size = 6) +
transition_reveal(data_aplicacao) +
scale_color_viridis_d() +
theme_bw(20) +
shadow_wake(wake_length = 0.5, size = 3) +
ease_aes("cubic-in") +
labs(x = "Data de referência", y= "% da população vacinado com a 2a dose",
titulo = "Evolução da vacinação contra COVID-19 no Brasil por região",
caption = "Fonte: openDataSus/Base dos dados")
animate(grafico_por_regiao, height = 600, width =800)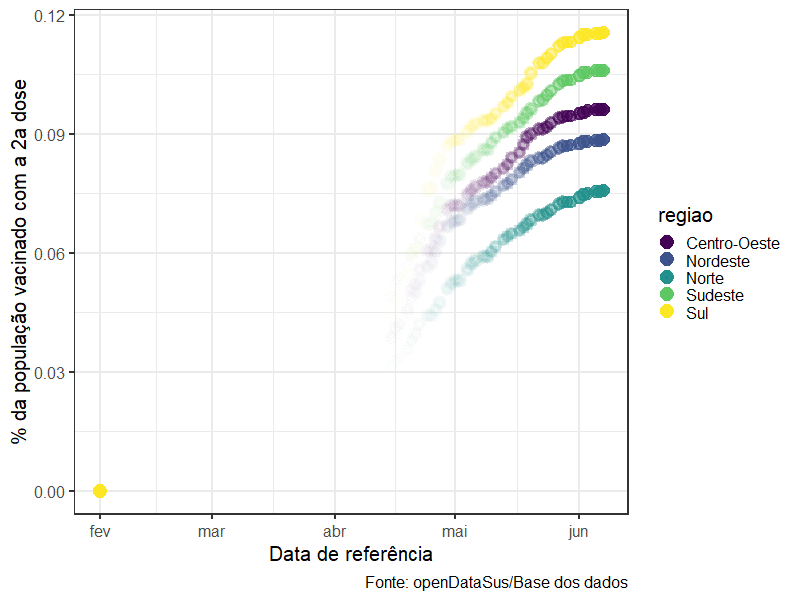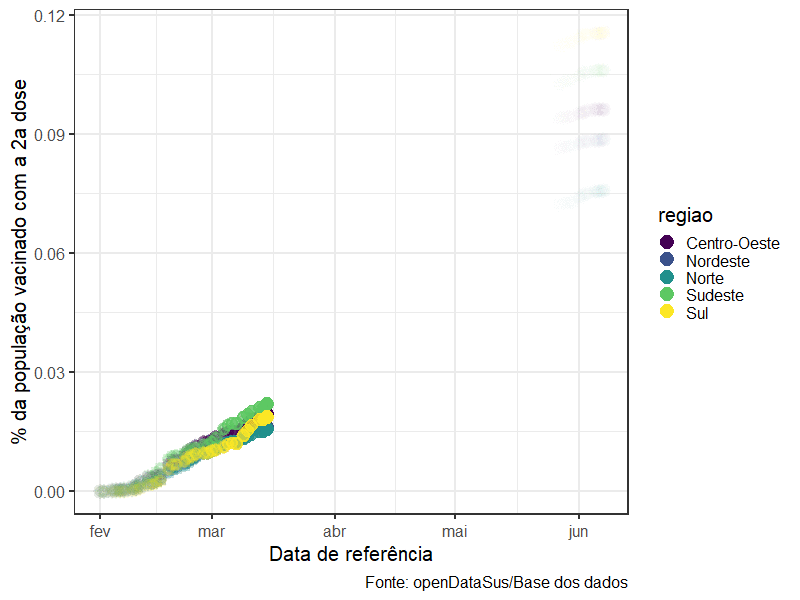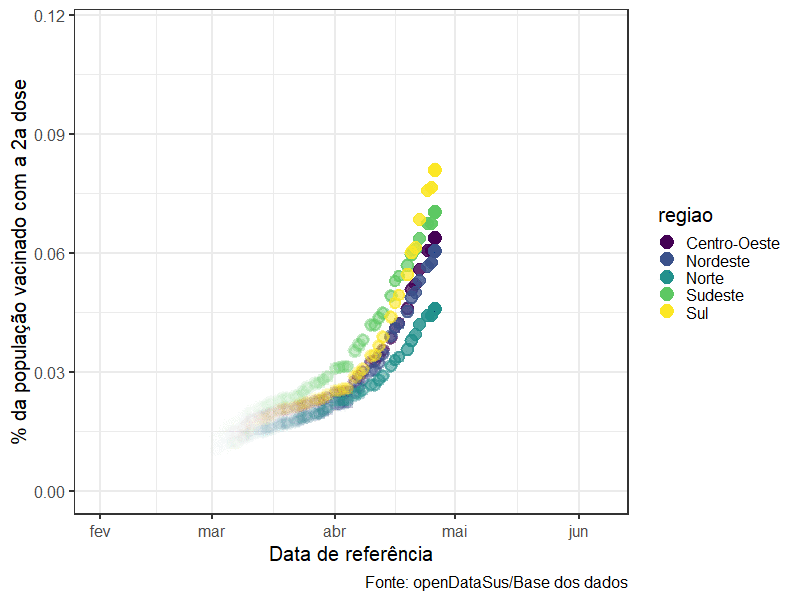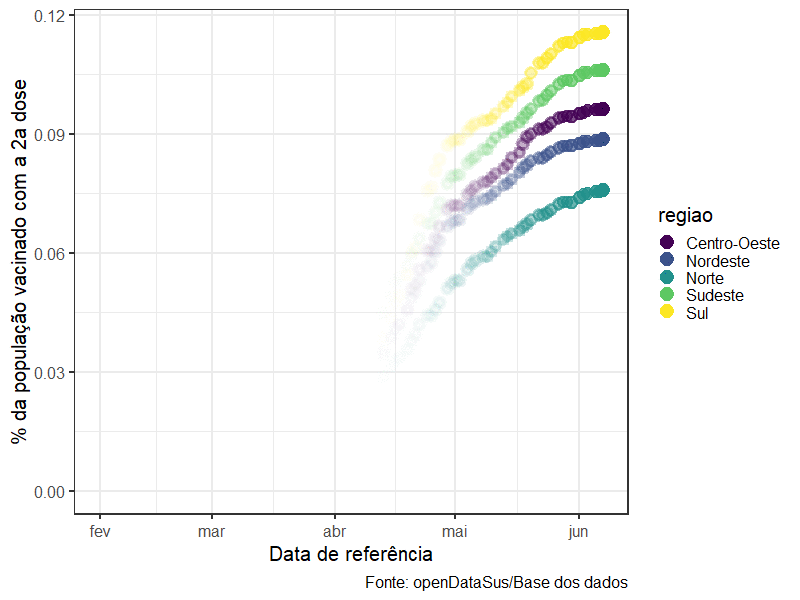
Gostou? Quer saber mais?
Confira a nossa live!
Se você quiser aprender um pouco mais sobre RMarkdown e Visualização de Dados em geral, dê uma olhada no nosso curso Relatórios e visualização de dados e aproveite!


