O que você vai aprender a fazer ao final da leitura é:
Ou seja, uma vez que você tenha um modelo de XGBoost ajustado dentro do seu R, dá para pedir para o {tidypredict} gerar a query de SQL que calcularia o score. PS: o XGBoost do gif tem 2 árvores.
Motivos para usar
Se existir um servidor com SQL muito poderoso (ou cluster, etc), a predição pode ser feita diretamente dentro do banco de dados, sem necessidade de outras ferramentas. Além disso, colocar em produção estará a um CTRL+C/CTRL+V de distância =P.
Preditor do XGBoost
O valor predito pelo XGBoost é, no fundo, uma soma de CASE WHEN’s (ou IF ELSE’s). O número de árvores que vai definir quantos CASE WHEN’s serão somados.
A cara do preditor de um XGBoost com 2 árvores ficaria assim no SQL:
<SQL> 0.0 + CASE
WHEN ((`dose` < 5.0 OR ((`dose`) IS NULL))) THEN (-1.95000005)
WHEN (`dose` >= 14.0 AND `dose` >= 5.0) THEN (-1.6500001)
WHEN ((`dose` < 10.0 OR ((`dose`) IS NULL)) AND (`dose` < 14.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (1.05000007)
WHEN (`dose` >= 10.0 AND (`dose` < 14.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (1.35000002)
END + CASE
WHEN ((`dose` < 5.0 OR ((`dose`) IS NULL))) THEN (-1.36500013)
WHEN (`dose` >= 14.0 AND `dose` >= 5.0) THEN (-1.15499997)
WHEN ((`dose` < 10.0 OR ((`dose`) IS NULL)) AND (`dose` < 14.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (0.734999955)
WHEN (`dose` >= 10.0 AND (`dose` < 14.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (0.945000052)
END + 0.5Código minimal
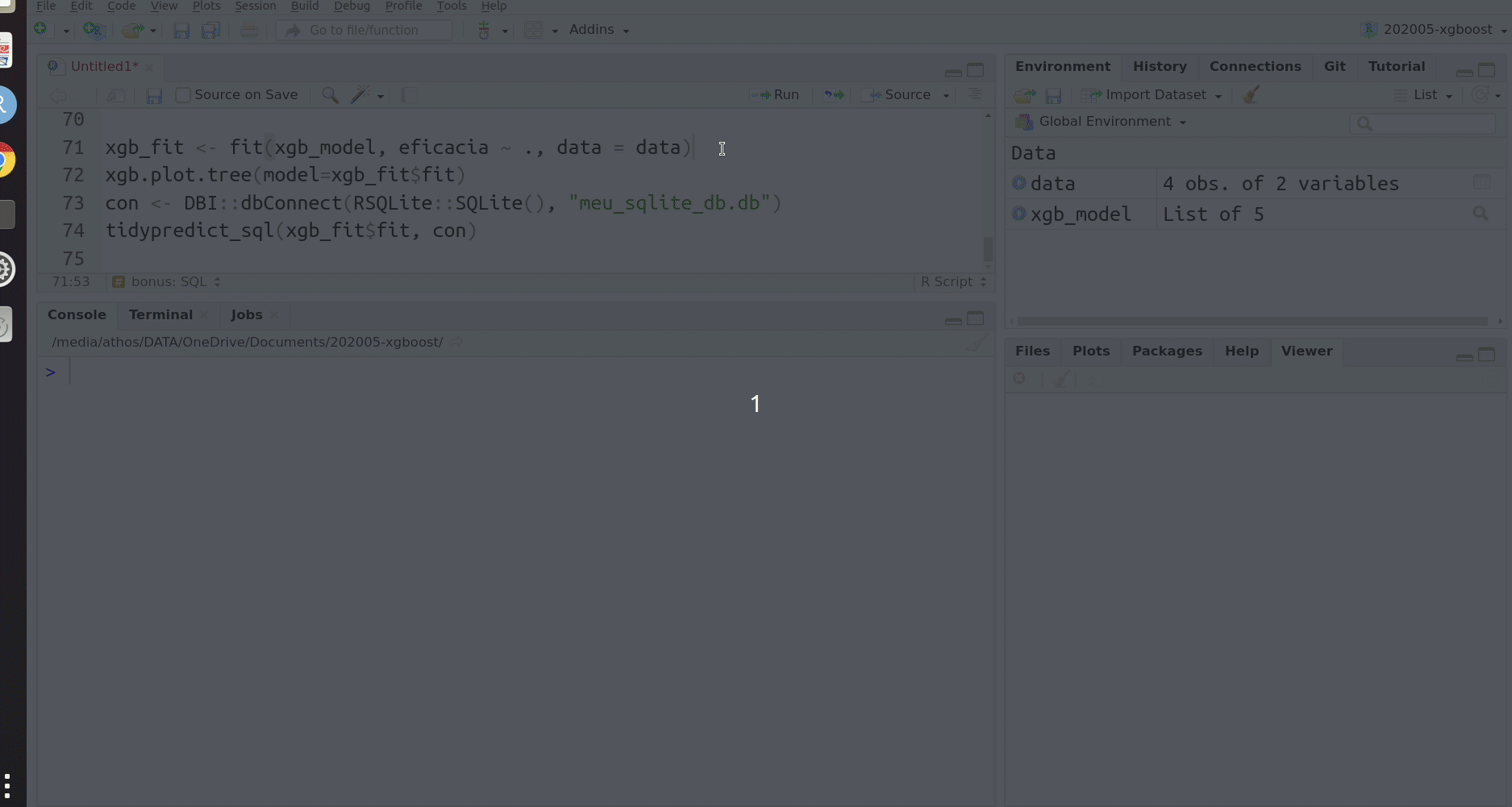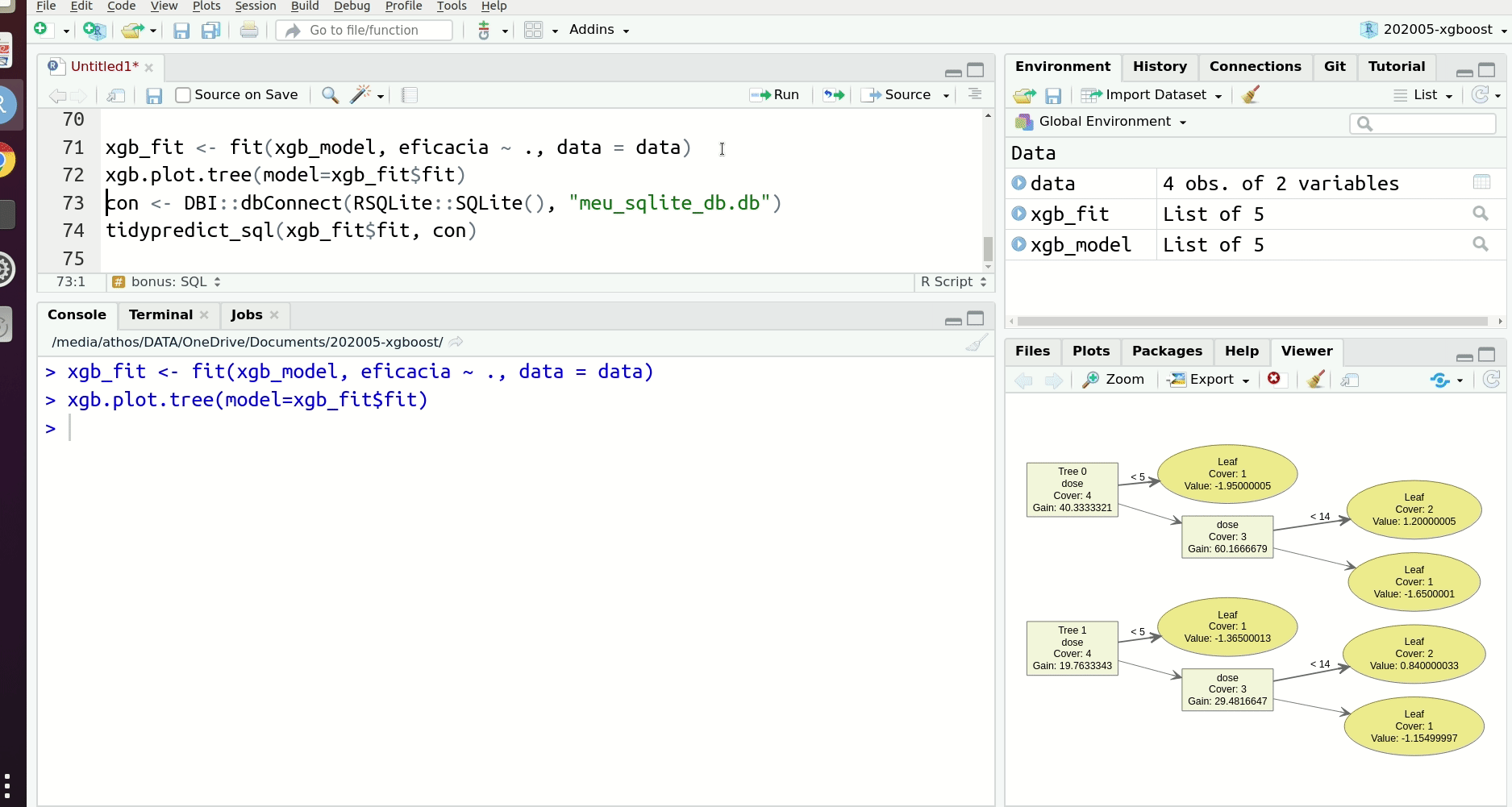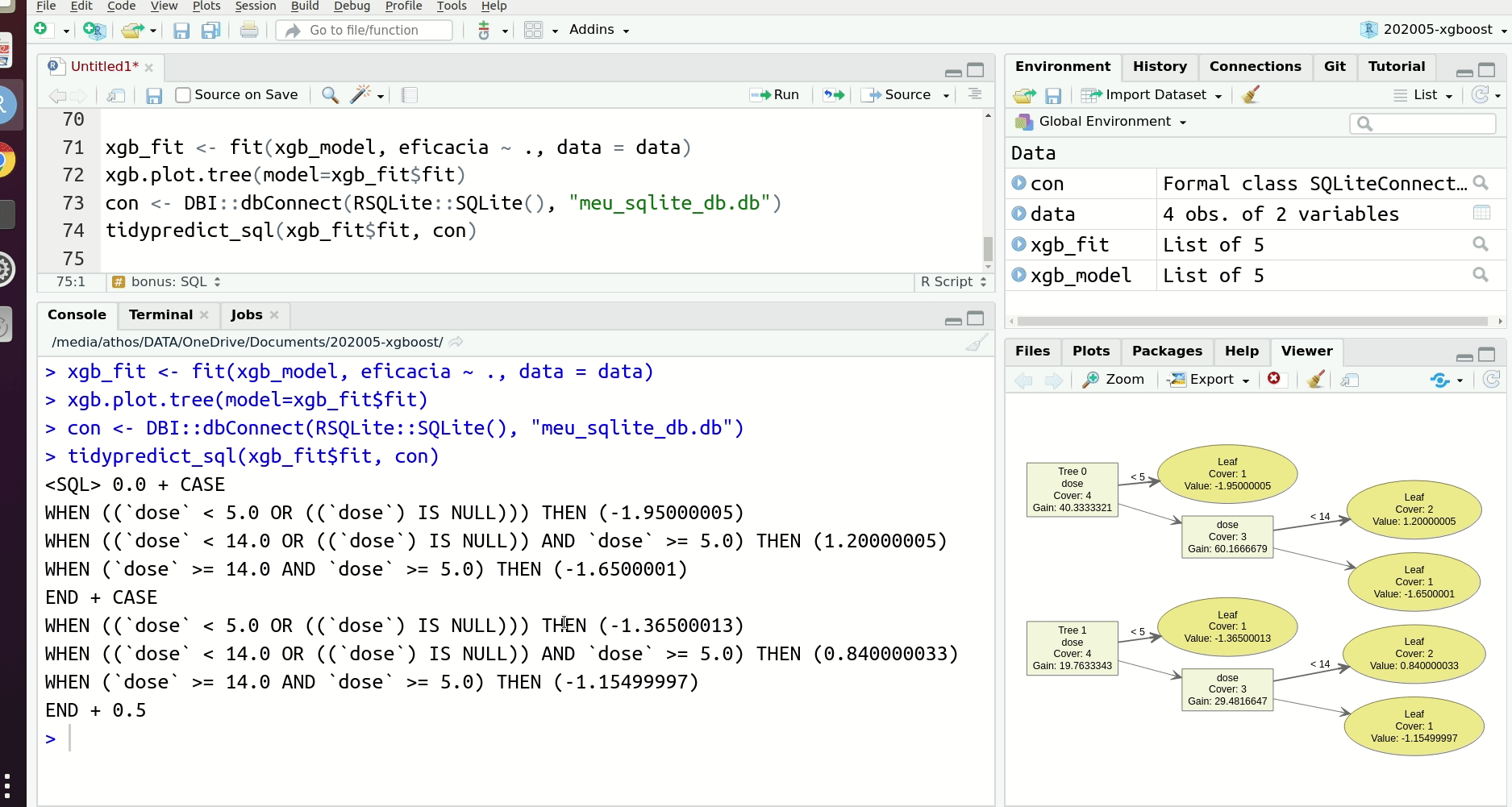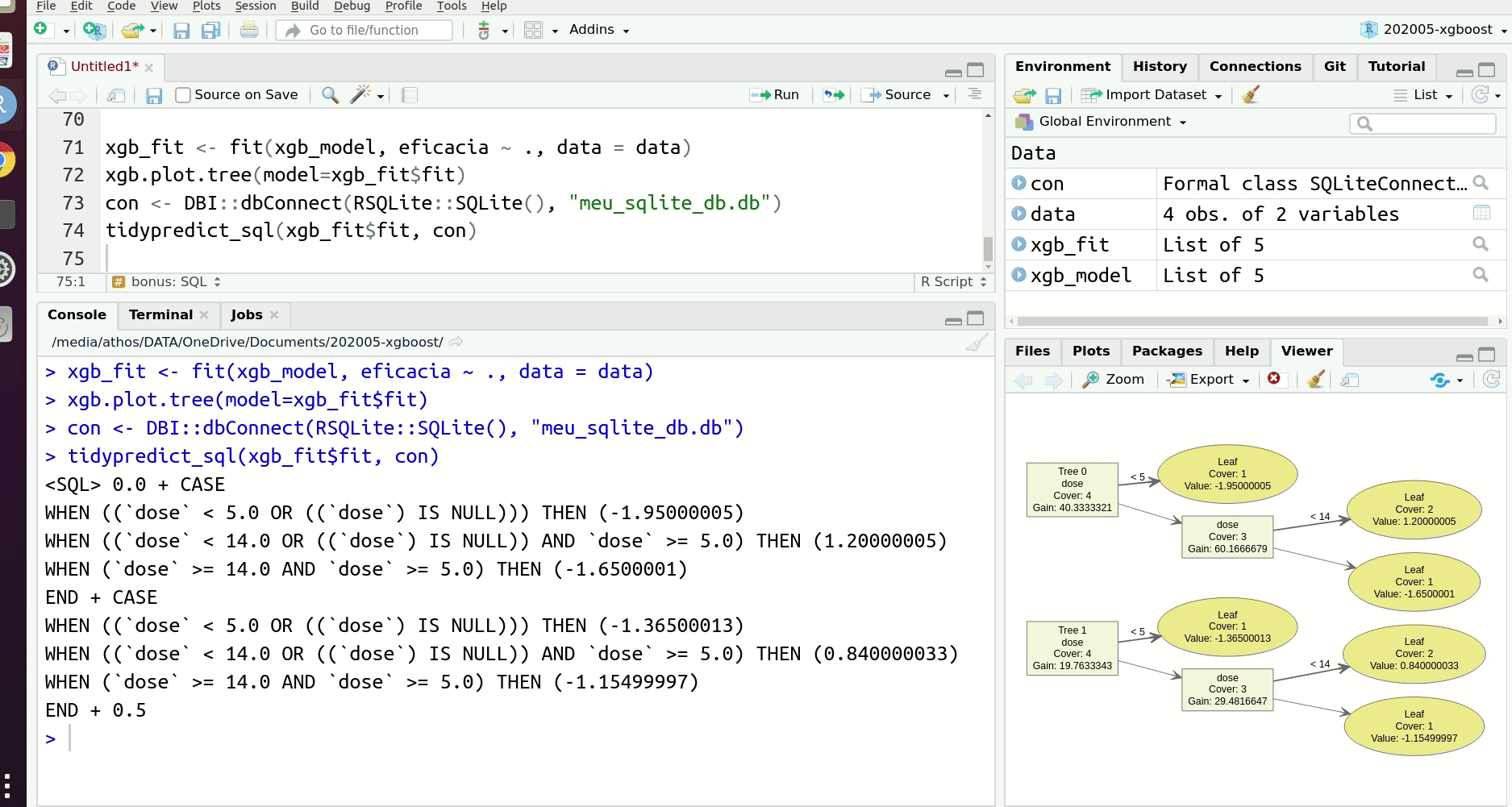
O verdadeiro herói do código abaixo é a linha tidypredict_sql(xgb_fit$fit, con) do pacote {tidypredict} que recebe o objeto com o XGBoost ajustado e retorna a query de SQL com os CASE WHEN’s.
Ajuste do XGBoost com {tidymodels}
library(tidymodels)
library(tidypredict)
library(xgboost)
# dados ----------------------------------------------------
data <- tibble(
dose = c(2, 8, 12, 18),
eficacia = c(-6, 4, 5, -5)
)
# especificacao do modelo ---------------------------------
xgb_model <- boost_tree(min_n = 1,
trees = 2
) %>%
set_engine("xgboost", lambda = 0) %>%
set_mode("regression")
# ajuste do modelo -----------------------------------------
xgb_fit <- fit(xgb_model, eficacia ~ dose, data = data)
# Conexão com o banco SQL ----------------------------------
con <- DBI::dbConnect(RSQLite::SQLite(), "meu_sqlite_db.db")
# Transcrevendo XGBoost para SQL ---------------------------
tidypredict_sql(xgb_fit$fit, con)
## <SQL> 0.0 + CASE
## WHEN ((`dose` < 5.0 OR ((`dose`) IS NULL))) THEN (-1.95000005)
## WHEN (`dose` >= 15.0 AND `dose` >= 5.0) THEN (-1.6500001)
## WHEN ((`dose` < 10.0 OR ((`dose`) IS NULL)) AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (1.05000007)
## WHEN (`dose` >= 10.0 AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (1.35000002)
## END + CASE
## WHEN ((`dose` < 5.0 OR ((`dose`) IS NULL))) THEN (-1.36500013)
## WHEN (`dose` >= 15.0 AND `dose` >= 5.0) THEN (-1.15499997)
## WHEN ((`dose` < 10.0 OR ((`dose`) IS NULL)) AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (0.734999955)
## WHEN (`dose` >= 10.0 AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (0.945000052)
## END + 0.5Usando o {dplyr} para rodar código no SQL
# apenas subindo os dados no banco de dados SQL ------
copy_to(con, data, "data", overwrite = TRUE)
# Criando coluna `pred` com as predições em SQL ------
data_sql <- tbl(con, "data") %>%
mutate(
pred = !!tidypredict_sql(xgb_fit$fit, con)
)
# resultado -------------------------------------------
data_sql
## # Source: lazy query [?? x 3]
## # Database: sqlite 3.35.5 [D:\blog\content\posts\meu_sqlite_db.db]
## dose eficacia pred
## <dbl> <dbl> <dbl>
## 1 2 -6 -2.82
## 2 8 4 2.29
## 3 12 5 2.80
## 4 18 -5 -2.31PS: a função tidypredict_sql() devolve uma string e por isso usamos o operador !! para interpretar como código de R literal. Essa parte pode ser confusa porque o dplyr vai pegar o código, traduzir para o literal, mas não vai rodar o código no R, vai traduzir do R para o SQL diretamente (e é por isso que dá certo porque a string não é um código de R válido =P).
SQL por trás dos panos
A tabela data_sql gerada acima pelo {dplyr} é, na verdade, uma query SQL. A função show_query() mostra a tradução resultante:
show_query(data_sql)
## <SQL>
## SELECT `dose`, `eficacia`, 0.0 + CASE
## WHEN ((`dose` < 5.0 OR ((`dose`) IS NULL))) THEN (-1.95000005)
## WHEN (`dose` >= 15.0 AND `dose` >= 5.0) THEN (-1.6500001)
## WHEN ((`dose` < 10.0 OR ((`dose`) IS NULL)) AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (1.05000007)
## WHEN (`dose` >= 10.0 AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (1.35000002)
## END + CASE
## WHEN ((`dose` < 5.0 OR ((`dose`) IS NULL))) THEN (-1.36500013)
## WHEN (`dose` >= 15.0 AND `dose` >= 5.0) THEN (-1.15499997)
## WHEN ((`dose` < 10.0 OR ((`dose`) IS NULL)) AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (0.734999955)
## WHEN (`dose` >= 10.0 AND (`dose` < 15.0 OR ((`dose`) IS NULL)) AND `dose` >= 5.0) THEN (0.945000052)
## END + 0.5 AS `pred`
## FROM `data`Resumo
- XGBoost para SQL é simples como uma linha de código:
tidypredict_sql(xgb_fit$fit, con) - A query SQL pode ser usada dentro do
mutate()com o!! - Predições com XGBoost não é nenhum bicho de sete cabeças, são singelas somas de CASE WHEN’s.
Apêndice: as duas árvores
library(DiagrammeR)
xgb.plot.tree(model=xgb_fit$fit)

