Salvar data.frames para ler depois é uma tarefa muito comum para quem trabalha com R, principalmente quando o seu processo possui algumas etapas mais demoradas e você não quer ter que rodar tudo de novo.
Veja aqui 3 formas fáceis, e rápidas para salvar o seu banco de dados e não perder tempo lendo novamente.
saveRDS
Talvez a função mais conhecida para salvar objetos do R. Ela salva em um formato binário que só pode ser lido pelo R. Por padrão comprime o arquivo após salvar, o que economiza espaço no disco, mas pode fazê-la levar mais tempo para rodar.
Considere um data.frame gerado pelo código abaixo:
nrOfRows <- 1e7
x <- data.frame(
Integers = 1:nrOfRows, # integer
Logicals = sample(c(TRUE, FALSE, NA), nrOfRows, replace = TRUE), # logical
Text = factor(sample(state.name, nrOfRows, replace = TRUE)), # text
Numericals = runif(nrOfRows, 0.0, 100), # numericals
stringsAsFactors = FALSE)Agora veja o tempo que demoramos para salvá-lo com o saveRDS.
system.time({
saveRDS(x, "~/Desktop/saveRDS.rds")
})
# user system elapsed
# 19.300 0.112 19.386O espaço ocupado pelo arquivo é de 95MB. Indicando para a função que você não deseja comprimir:
system.time({
saveRDS(x, "~/Desktop/saveRDS2.rds", compress = FALSE)
})
# user system elapsed
# 0.260 0.116 0.377O tempo vai para menos de 1s. Mas agora o arquivo ficou com 200MB.
O pacote readr tem uma função chamada write_rds que é um wrapper de saveRDS
que por padrão não comprime os arquivos, já que o Hadley diz que armazenamento é,
em geral, muito mais barato do que tempo de processamento.
É importante também verificar o tempo que demoramos para ler o arquivo novamente para o R. No caso ler o arquivo comprimido demora 2x mais do que o arquivo não comprimido.
system.time({
a <- readRDS("~/Desktop/saveRDS.rds")
})
# user system elapsed
# 1.068 0.040 1.105
system.time({
a <- readRDS("~/Desktop/saveRDS2.rds")
})
# user system elapsed
# 0.380 0.068 0.447Para salvar data.frames do R no disco, saveRDS é sempre a minha primeira opção:
é relativamente rápido para ler e escrever e não exige instalação de nenhum pacote.
Além disso, o saveRDS serve para praticamente qualquer tipo de objeto do R, ou seja,
você pode usá-lo para salvar os modelos que você ajustou ou qualquer outra coisa.
As principais desvantagens dessa função para as outras que mostrarei a seguir são:
- só pode ser lido pelo R
- não permite que você leia apenas um subset das linhas/ colunas.
feather
feather é um formato de arquivo desenvolvido por duas pessoas muito fodas. Wes McKinney,
autor do Pandas (principal biblioteca de manipulação de dados do python) e Hadley Wickham,
principal desenvolvedor do tidyverse.
O feather é bem rápido para salvar data.frames no disco, tempo comparável a
salvar o arquivo sem comprimir usando o saveRDS. Mas só isso não é o suficiente para
ser necessário usá-lo, já que neste caso o saveRDS rápido o suficiente.
A principal vantagem do feather é que ele foi criado para ser um formato de
compartilhamento de data.frames entre diversas linguagens de programação. Existem
pacotes para ler arquivos .feather escritos em R, python, Julia: as três principais
linguagens para análise de dados.
O feather também permite que você leia apenas algumas linhas ou colunas do dataset, o
que muitas vezes é útil para fazer consultas mais rápidas na base sem ter que ler tudo para
a RAM de uma vez só.
library(feather)
system.time({
write_feather(x, "~/Desktop/feather.feather")
})
# user system elapsed
# 0.172 0.084 0.253O arquivo produzido pesa 162MB. Para ler o arquivo salvo:
system.time({
a <- read_feather("~/Desktop/feather.feather")
})
# user system elapsed
# 0.112 0.020 0.131Usando o feather para ler apenas algumas linhas e colunas.
a <- feather("~/Desktop/feather.feather")
b <- a[5000:6000, 1:3]fst
fst é um pacote para ler e escrever data.frames de forma muito rápida.
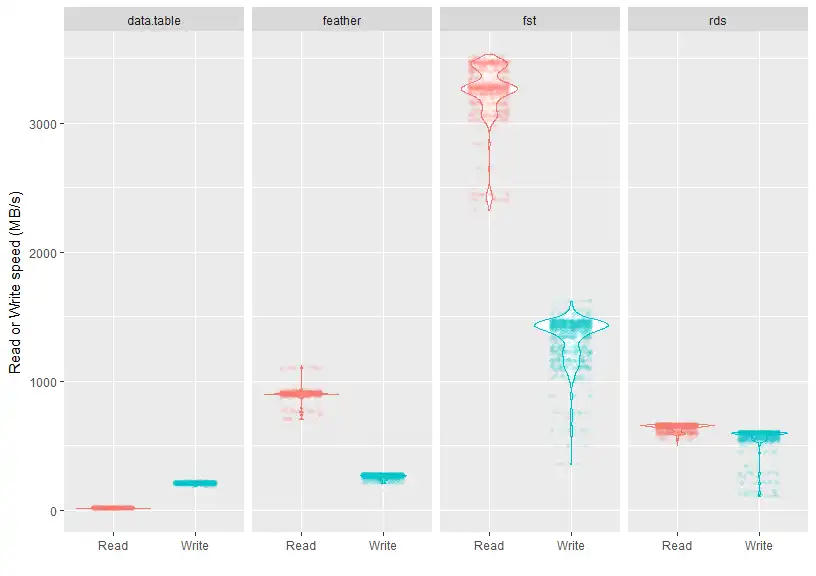
A imagem (retirada daqui) acima mostra a sua velocidade. O fst é mais ou menos 3 vezes mais rápido
para ler os arquivos salvos e cerca de 2x mais rápido para escrevê-los. O arquivo
salvo pelo fst é também um pouco menor: 130MB.
Ler e escrever é, assim como as outras opções, tão simples como usar uma função:
library(fst)
# salvar
write.fst(x, "/home/daniel/Desktop/dataset.fst")
# ler
a <- read.fst("~/Desktop/dataset.fst")
# ler apenas algumas linhas e colunas
b <- read.fst("~/Desktop/dataset.fst", c("Logicals", "Text"), 2000, 4990)Note Como o Sillas mencionou nos comentários, a versão do CRAN do fst salva
datas como numericos no arquivo. Os números podedm sser convertidos para data novamente
usando a função as.Date do pacote zoo, mas tem que tomar cuidado!
Conclusão
Use sempre
saveRDSereadRDS, se precisar de velocidade, salve com o argumentocompress = FALSEpara não comprimir o arquivo.Se você for ler a base em python ou Julia e quiser um formato padronizado, use o
feather.Se você for realmente ler e escrever os seus dados muitas vezes e você precisar de velocidade, use o
fst.


